Opprettelse av Genos historiske avlsprogram i en digital tvilling
Denne artikkelen er del to av en serie i tre deler om bruk av «digital tvilling» i Genos avlsprogram. Den digitale tvillingen er en simulering som er designet for å være så lik sin virkelige motpart som mulig.
Avlsforsker i Geno
janez.jenko@geno.no
Dette er del 2 av en artikkelserie som skal omhandle:
Del 1: Prosjektet og potensialet til digital tvilling i praksis
Del 2: Opprettelse av Genos historiske avlsprogram
Del 3: Testing av fremtidige alternativer i Genos avlsprogram
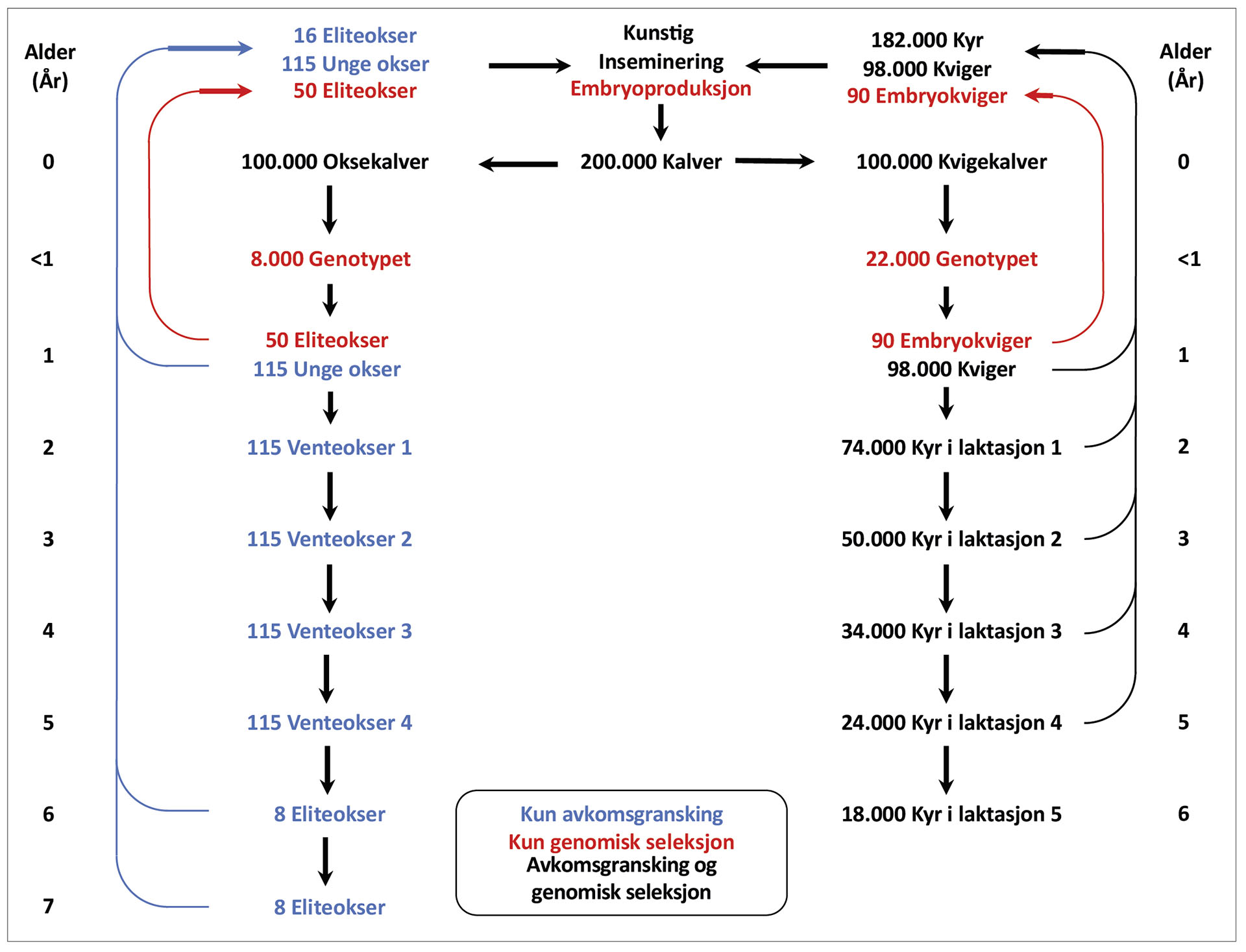
Figur. Avkomsgransking og genomisk seleksjon i Genos historiske avlsprogram integrert i digital tvilling
I den første artikkelen, som ble publisert i Buskap 5/2023, introduserte vi den digitale tvillingen og diskuterte potensialet for praktisk anvendelse av denne teknologien. I denne artikkelen skal vi snakke om opprettelsen av den digitale tvillingen av Genos historiske avlsprogram, som dekker perioden fra begynnelsen og fram til i dag.
Fra opprinnelsen til avkomsgransking
Vi begynte med å simulere det komplette storfegenomet, som omfatter 29 autosomale kromosomer, og sporet den demografiske modellen for storfe tilbake til opprinnelsen, inkludert domestisering og raseutvikling. Derfra simulerte vi 45 år med avkomsgransking, og innlemmet stamtavleinformasjon for å estimere avlsverdier. Hvert år valgte vi ut 115 ungokser basert på det beste foreldregjennomsnittet. Når oksene ble ett år gamle, ble de brukt til inseminasjon på 40 prosent av NRF-kyrne. Som et resultat hadde hver ungokse nesten 700 avkom da de ble to år gamle, med like andeler av okse- og kvigekalver. Om lag 250 av kvigekalvene ble senere kyr som fullførte sin første laktasjon. Fenotypedataene som ble samlet inn i løpet av denne laktasjonsperioden, ble deretter brukt til å estimere avlsverdiene til deres fedre, som allerede var fem år gamle på det tidspunktet. Vi valgte ut de åtte oksene med høyest avlsverdier fra gruppen av 115 avkomsgranskede okser. Disse ble valgt ut som eliteokser for de neste to årene. Som et resultat ble 16 eliteokser brukt til å inseminere 60 prosent av kupopulasjonen hvert år.
Genomisk seleksjon
Etter 45 år med avkomsgransking, simulerte vi fem år med genomisk seleksjon. Først genotypet vi oksene som var avkomsgransket i de foregående 40 årene med avkomsgransing, for å etablere en referansepopulasjon for genomisk seleksjon. Deretter genotypet vi årlig 8 000 oksekalver og 22 000 kvigekalver. De 50 beste oksekalvene ble valgt ut som eliteokser for inseminering av kyr, mens 90 kvigekalver ble valgt ut for embryooverføring. Hver kvige produserte 20 embryoer og ble deretter gjeninnført drektige i kupopulasjonen. Genotypede dyr ble inkludert i referansepopulasjonen når deres fenotypiske data ble tilgjengelige. Dette ble muliggjort gjennom implementeringen av en ett-stegstilnærming for prediksjon av avlsverdier, lignende den som brukes i Genos avlsprogram.
Endringer på oksesiden
Betydelige endringer skjedde på oksesiden av seleksjonsprosessen med innføringen av genomisk seleksjon (se figur). Venteokser var ikke lenger til stede, da GS- okser oppnådde eliteoksestatus i svært ung alder. På kvigesiden forble seleksjonsordningen relativt uendret, selv om innføringen av embryokviger la til en ny dimensjon. Imidlertid var kjernen i programmet fortsatt avhengig av kupopulasjonen som produserte melk på gårdene. Vi simulerte kyr i flere laktasjoner med varierende utskiftningsrater, beregnet ut fra NRF-populasjonen. Alle disse kyrne var en del av kjernen og representerte potensielle framtidige oksemødre.
Sammenligning med Genos avlsprogram
Den digitale tvillingen omfatter ikke alle historiske endringer i Genos avlsprogram, men fokuserer bare på de som har hatt størst innvirkning på dagens NRF-populasjon. For å sikre at den digitale tvillingen er tett i samsvar med virkeligheten, sammenlignet vi utviklingen i genetisk fremgang og innavlsnivå i de siste 50 årene med NRF-avl, med den i den digitale tvillingen. Avvik ble observert. Vi så spesielt høyere nivåer av innavl i den digitale tvillingen. Denne forskjellen skyldtes sannsynligvis import av dyr fra andre rasepopulasjoner, som ikke ble tatt hensyn til i den digitale tvillingen. For å rette opp dette, justerte vi valget av eliteokser og embryokviger for å favorisere seleksjon innen familie. Vi endret også valget av SNPer som ble brukt for genotyping i den digitale tvillingen, for å matche frekvensen av minoritetsallelene som finnes i NRF-populasjonen.
Fundament for å teste alternative strategier
Det historiske avlsprogrammet fungerer som et fundament for å teste alternative strategier i det fremtidige avlsprogrammet. Her kan vi utforske ulike tilnærminger for genotyping og fenotyping, sammen med ulike seleksjonsstrategier, som alle påvirker fremtidig genetisk forbedring av NRF. Mer informasjon kommer i neste nummer av Buskap. Der skal vi gå nærmere inn på effektene av ulike antall fenotyper på genetisk fremgang.



2.png)