Funn av mutasjon som gjev emryodød og omløp i NRF
BTA8H blir no implementert i Geno avlsplan på samme måte som fruktbarhetsdelesjonen og AH1.
arne.gjuvsland@geno.no
Begge avlsforskere i Geno
janez.jenko@geno.no

Frekvensen av bærarar av BTA8H svinga rundt 10 prosent i perioden frå 1980-2000, men seinare har frekvensen gått litt ned slik at den er rundt 6 prosent i dagens NRF-populasjon. Foto: Rasmus Lang-Ree
Ved genotyping av dyr blir det kontrollert om dyret er bærar av kjente mutasjonar med negativ effekt på fruktbarheit og overlevelse slik som fruktbarhetsdelesjonen på kromosom 12 og AH1. Dette er såkalte recessive letale mutasjonar, som betyr at avkom som har fått genvarianten frå begge foreldre dør enten som embryo, seinare i fosterutviklinga eller er dødfødde. Dyr som har ein kopi av mutasjonen blir kalla bærarar og blir friske og gode produksjonsdyr. Bærarar vil også ha normal fruktbarheit, med mindre dei blir inseminert med bærar av samme recessive letale mutasjon.
Avvik frå mendelsk nedarving
Mendel sine kryssingsforsøk avdekka reglar for nedarving hos planter og dyr. Desse viser seg i praksis ved at genotypane til avkom fordeler seg etter faste forholdstal bestemt av genotypane til foreldra.
Far |
Mor |
|||
|---|---|---|---|---|
Genotype foreldre |
AB |
AB |
||
Kjønnceller |
A |
B |
A |
B |
Genotypar avkom |
AA AB AB BB |
|||
Tabellen viser eit eksempel på dette for eit enkelt gen med to genvariantar A og B, i familiar der både mor og far har genotypen AB. Dersom ein ser på genotypane til mange kalvar etter slike foreldre vil dei fordela seg etter forholdstala 1(AA):2(AB):1(BB). Men, dersom B-varianten er recessiv letal vil dei inseminasjonane som gir BB enda med omløp, kasting eller dødfødsel og ingen levande kalvar vil vera homozygote BB. Blant genotypa kalvar vil forholdstala derfor vera 1(AA):2(AB):0(BB). Denne mangelen på homozygotarer er ein signatur ved recessive letale mutasjonar, og dersom ein har genotypar frå mange dyr kan dette brukast til å finna slike mutasjonar.
Analyse av genotypar og kukontrolldata
Hausten 2020 gjorde me eit søk etter recessive letale mutasjonar i genotypedata frå 45 000 SNPar (genetiske markørar på chipen som blir brukt til genotyping) i
113 000 NRF-dyr. Me delte kromosoma inn i regionar og innanfor kvar region splitta me opp genotypen til kvart dyr i to delar. Desse delane blir kalla haplotypar og tilsvarer bidraga frå far og mor til genotypen til dyret. Så leita me etter haplotypar som fins i mange individ, men der ingen av individa er homozygote for haplotypen. Dei haplotypane som viste dette mønsteret følgde me opp med analyse av registrerte inseminasjonar og kalvingar frå Kukontrollen. Inseminasjonar der både okse og far til ku er bærar av ein recessiv letal mutasjon vil ha auka risiko for omløp, kasting eller dødfødslar. Når me kombinerte resultata frå genotypedata med Kukontrolldata stod me igjen med to haplotypar med tydeleg effekt på omløp. Den eine haplotypen er på kromosom 12 og er knytta til fruktbarhetsdelesjonen som er kjent frå før. Den andre er på kromosom 8 i eit område der ingen har rapportert om recessive letale mutasjonar tidlegare.
BTA8H – ein recessiv letal haplotype på kromosom 8
Den nyoppdaga haplotypen, som me kallar BTA8H, ligg omtrent 70 millionar basepar inn på kromosom 8, og den dekkar eit område på rundt 1,1 millionar basepar. BTA8H kan sporast tilbake i NRF-populasjonen til dei eldste genotypa oksane frå 1970-talet. Frekvensen av bærarar svinga rundt 10 prosent i perioden frå 1980-2000, men seinare har frekvensen gått litt ned slik at den er rundt 6 prosent i dagens NRF-populasjon. Eksempel på bærarar som har vore viktige i NRF-avlen er 10617 Skei, 10909 Tangvoll, 10115 Raastad, 5848 Øygarden, 4948 Brandstadmoen, 4939 Rånes og 3927 K.Reime.
I perioden 2004-2018 er det registrert 3,9 millionar inseminasjonar der både far til ku og seminoksen som er brukt er genotypa. Rundt 27 500 (ca. 0,7 prosent) av desse er inseminasjonar der både far til ku og seminokse er bærarar av BTA8H. Desse bærar-bærar inseminasjonane gjer meir omløp enn inseminasjonar der berre ein eller ingen av oksane er bærar (Figur 1). Figuren viser at BTA8H gjer lågare ikkje-omløpsgrad enn fruktbarheitsdelesjonen ved 35 og 56 dagar, men etter 150 dagar er ikkje-omløpsgraden lik. Dette tyder på at BTA8H gjer rask embryodød og tidleg omløp, mens fruktbarheitsdelesjonen gjer seine omløp eller kasting.
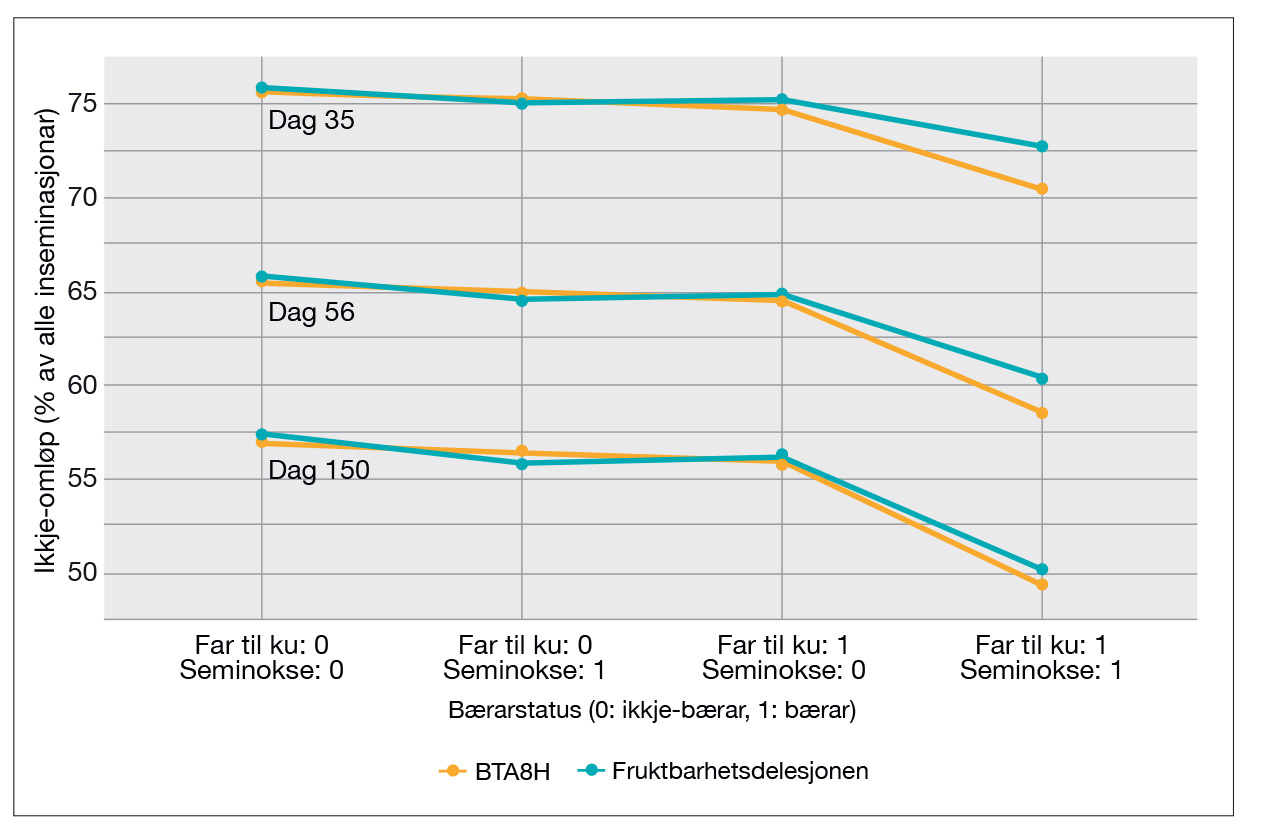
Figur 1: Ikkje-omløpsprosent for ca. 3,9 millionar inseminasjonar i perioden 2004-2018 der både far til ku og seminokse er genotypa.Inseminasjonane er gruppert etter bærarstatus. Ca. 3,2 millionar er inseminasjonar der verken far til ku eller seminoksen bærarar (x-akse: Far til ku: 0 Seminokse: 0). I ca. 317 000 inseminasjonar er berre seminoksen bærar og rundt 339 000 er berre far til ku bærar, mens ca. 27 500 er inseminasjonar der både far til ku og seminokse er bærarar (x-akse: Far til ku: 1 Seminokse: 1).
Implementering i Geno Avlsplan og avlsprogrammet
BTA8H blir no implementert i Geno avlsplan på samme måte som fruktbarhetsdelesjonen og AH1. Genotypa dyr vil få bestemt bærarstatus og dersom kua er bærar vil avlsplanen unngå oksar som også er bærar. Det er desse bærar-bærar inseminasjonane som har auka risiko for omløp og er viktige å unngå. Med den frekvensen BTA8H har i NRF i dag vil under 0,5 prosent av inseminasjonane vera bærar-bærar inseminasjonar så effekten på omløp over heile NRF-populasjonen er svært liten. Dei viktigaste tiltaket er derfor å unngå bærar-bærarinseminasjonar og avlsplanen vil sørga for dette, det er ikkje tilrådd å utrangera dyr på grunn av bærarstatus. I avlsprogrammet vil me overvåka frekvensen og sørga for at den ikkje aukar. BTA8H-status vil også bli tatt med i vurderinga av seleksjon av embryokviger og eliteoksar, men bærarar med høg avlsverdi eller god profil på indeksane vil fremdeles bli selektert.
Vidare arbeid med finkartlegging
Haplotypen BTA8H er assosiert med ein recessiv letal mutasjon, haplotypen har stor bruksverdi fordi den kan brukast til å indentifisera bærarar, men den underliggande såkalte kausale mutasjonen som er årsaka til embryodød og genet eller gena som er involvert er framleis ukjente. Chipen som blir brukt til genotyping har 26 SNPar i dette området på kromosom 8, men i sekvensdata av NRF-oksar fins det nesten 6 000 ulike mutasjonar i det samme området. Det er ein vanskelig leitejobb å finna den eine kausale mutasjonen, men me vil ta BTA8H inn i pågåande forskningsprosjekt der me alt arbeider med sekvensdata og bioinformatikk og fortsetta leitinga der.
Ordliste
Homozygot: Brukt om individ som har to like genvariantar (allel) av ein mutasjon.
Heterozygot: Brukt om individ som har to ulike genvariantar (allel) av ein mutasjon.
Haplotype: Halv-genotype tilsvarande genotypen til ein av kjønncellene som gav opphav til eit individ. Kroppsceller hos dyr er diploide, dei har to av kvart kromosom, eit frå far og eit frå mor. Ved vanlig genotyping og GS-testing blir genvariantane på begge kromosomkopiar slått sammen. Men, for andre typar analysar er det nødvendig å skilla ut bidraga frå far og mor ved å dela opp genotypane på eit kromosom eller kromosomområde i to haplotypar.



2.png)